RT-qPCR-eksperimento inkluzivas RNA-eltiron kaj kvalitan taksadon, inversan transskribon kaj qPCR tri paŝojn, ĉiu paŝo havas multajn antaŭzorgojn, ni enkondukos detale sube.
Ⅰ.Takso de kvalito de RNA
En RT-qPCR-eksperimento, post la kompletigo de RNA-ekstraktado, la kvalito de RNA devas esti taksita, kaj la sekva eksperimento povas esti farita nur post kiam ĝi estas kvalifikita.Taksadmetodoj inkludas spektrofotometron, Agilent-ĝelelektroforezon, Agilent 2100-analizon, inter kiuj la plej ofte uzita spektrofotometro kaj agaroseĝelelektroforezometododetekto.Oni devas rimarki, ke ĉi tiuj du metodoj devas esti uzataj kune por kompletigi la detekton kaj analizon de RNA-koncentriĝo, pureco kaj integreco, por certigi la kvaliton de RNA.
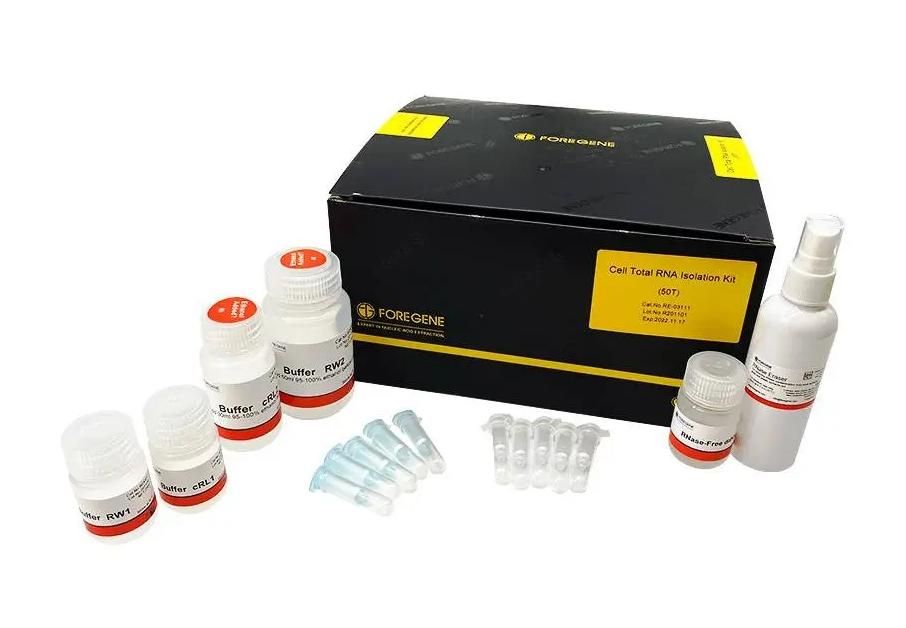
Rilata RNA-Izola Ilaro:
Tre purigita kaj altkvalita totala RNA povas esti akirita de diversaj kulturitaj ĉeloj en 11 min.
Rapide kaj efike ĉerpas altpuran kaj altkvalitan totalan RNA el diversaj bestaj histoj.
Spektrofotometro:
La spektrofotometro estas ĉefe uzata por determini la koncentriĝon kaj purecon de RNA, sed ĝi ne povas detekti la integrecon de RNA kaj genoma restaĵo.Inter ili, A260/280 kaj A260/230 estas gravaj parametroj por detekto de RNA-pureco, kaj RNA-pureco povas esti detektita laŭ la fluktuo de iliaj valoroj:
1. 1.9< A260/280< 2.1, indikante ke RNA-pureco estas bona;A260/280<1.9, indikante ke povas ekzisti proteinrestaĵo en RNA;A260/280>2.1, indikante eblan partan degeneron de RNA, kiu povas esti plue konfirmita per agaroseĝelektroforezo.
2. 2.0< A260/230< 2.2, indikante ke RNA-pureco estas bona;A260/230< 2.0, indikante ke povas ekzisti restaĵoj de organikaj reakciiloj en RNA, kiel fenoloj, etanolo aŭ sukeroj.
Elektroforezo de agaroza ĝelo:
Agarose-ĝela elektroforeza analizo povas analizi RNA-integrecon, genaron kaj proteinrestaĵojn, sed ne povas precize kvantigi la koncentriĝon de RNA aŭ detekti la restaĵojn de organikaj reakciiloj.Prenu eŭkariotajn RNA-ŝablonojn ekzemple:
1. La RNA estis submetita al elektroforezo de agaroza ĝelo.Se ekzistis nur tri ununuraj grupoj de 28sRNA, 18sRNA kaj 5.8sRNA sur la ĝelmapo, ĝi indikas ke la eltirita RNA estas sendifekta.Se ekzistas trena fenomeno, ĝi indikas partan degeneron de RNA.
2. Se estas ununura hela bando inter la glutruo kaj la 28sRNA-bendo, povas esti genoma DNA-restaĵo.
3. Se bandoj aperas en la glutruo, tio indikas, ke povas esti restaĵoj de proteino kaj aliaj makromolekulaj substancoj.
Ⅱ. Inversa transskribo
Post kiam RNA-ekstraktado estas finita, ĝi devas esti inversigita en cDNA por postaj eksperimentoj, do la inversiga paŝo estas esenca.Inversa transskribo estos lanĉita de la elekto de inversa transkriptazo kaj enkonduko:
Selektado de inversa transkriptazo:
La tipaj inversaj transkriptazoj inkludas AMV RTase kaj MMLV RTase.La RNase H de AMV RTase havas fortan agadon, mallongan sintezan longon, malaltan sintezan kvanton kaj bonan termikan stabilecon (42 ~ 55 ℃).La agado de RNase H de MMLV RTase estas malforta, la sintezolongo estas longa, la sinteza kvanto estas alta, kaj la termika stabileco estas malbona (37 ~ 42 ℃).
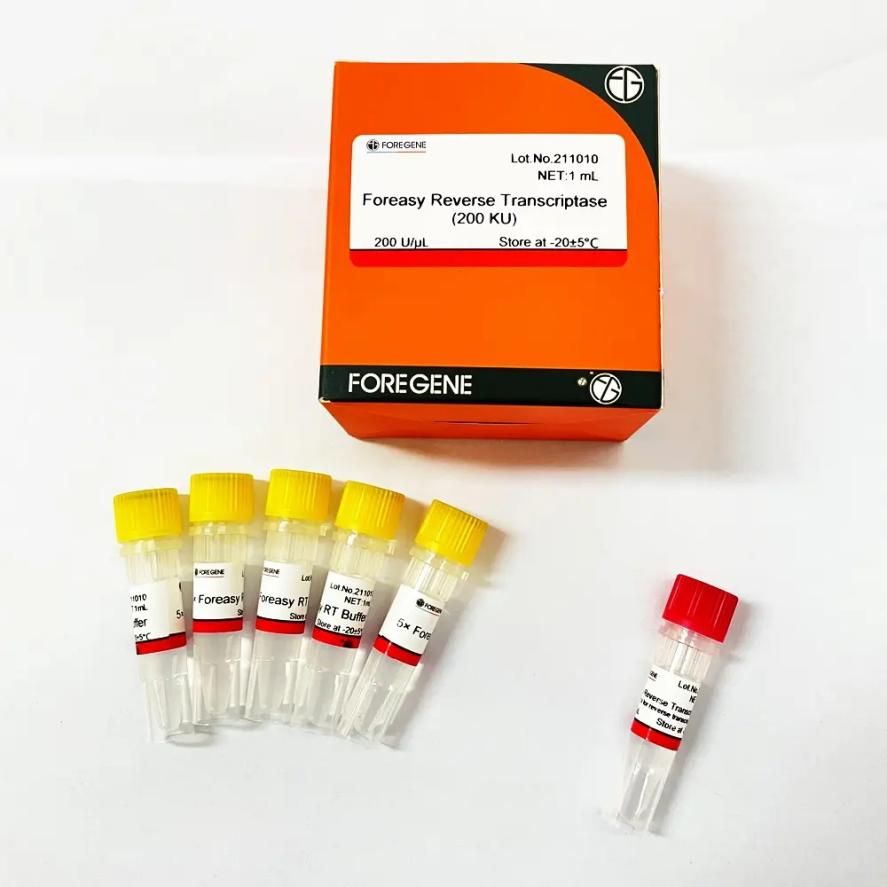
Ĉar RNase H-enzimo havas la funkcion de degradado de RNA-ŝablono, MMLV kun malforta RNase H-agado devus esti prefere elektita dum inversa transskribo, kaj post pli posta genetika inĝenierado, la termika stabileco de MMLV atingis kvalitan salton.Prenante ForegeneForeasy Reverse Transcriptase (M-MLV por inversa transskribo) ekzemple, ĝi estas nova inversa transkriptazo esprimita en E. coli realigita bakterioj uzanta genetikan rekombinigteknologion.Ĝi estas rekombina DNA-polimerazo kiu sintezas komplementan DNA-fadenon de unu-fadena RNA, DNA, aŭ RNA:DNA hibrido.Ĝi havas neniun RNase H-agadon, fortan stabilecon, fortan RNA-afinecon kaj altan detektan sentemon.
Foreasy Reverse Transcriptase (M-MLV por inversa transskribo)
Elekto de enkonduko:
Ĝenerale RT-enkondukoj falas en tri kategoriojn: oligo dT, hazardaj enkondukoj, kaj gen-specifaj enkondukoj.Elektu taŭgajn enkondukojn por uzo laŭ malsamaj eksperimentaj postuloj.
1. Se la ŝablono estas de eŭkariota origino kaj la malfrua cDNA estas uzata por rutina PCR plifortigo, Oligo (dT) estas rekomendita;Se la posta eksperimento estas nur uzita por qPCR, Oligo (dT) rekomendas esti miksita kun hazardaj enkondukoj por plibonigi la efikecon de inversa transskribo.
2. Se la ŝablono estas de prokariotoj, Hazardaj Enkondukoj aŭ genaj specifaj enkondukoj estu elektitaj por inversa transskribo.
Ⅲ.qPCR
Fluoreskeca kvantigo estas ĉefe ellaborita el la elekto de kvantaj metodoj, komencaj dezajnaj principoj, ROX-elekto, reagsistema agordo kaj reagkondiĉoj, ktp.
Elekto de kvantaj metodoj:
Kvantaj metodoj estas dividitaj en relativajn kvantajn metodojn kaj absolutajn kvantajn metodojn.Relativa kvantigo povas esti uzata por detekti la efikon de certaj kuracmetodoj sur gena esprimo, detekti la diferencon de gena esprimo en malsamaj tempoj kaj kompari la diferencon de gena esprimo en malsamaj histoj.Absoluta kvantigo povas detekti la kvanton de nuklea acido en la viruso ktp.Farante eksperimentojn, ni devas elekti la taŭgajn kvantajn metodojn laŭ niaj propraj eksperimentoj.
Unuaj dezajnaj principoj:
La dezajno de enkonduko por qPCR estas rekte rilatita al la plifortiga efikeco kaj produktospecifo.Tial ĝuste desegni bonajn enkondukojn estas la unua paŝo de sukcesa qPCR.En la dezajno de enkonduko, la sekvaj principoj devas esti atentitaj kiam vi renkontas la principon de konvencia enkonduka dezajno:
1. La longo de la celfragmento estas kontrolita inter 100 kaj 300 bp;
2. Kruc-ekson-dezajno por eviti la influon de genoma DNA;
3. La desegnitaj enkondukoj devas esti provitaj por plifortiga efikeco, kaj nur kiam la plifortiga efikeco atingas la normon (90-110%) ili povas esti uzataj por kvantaj eksperimentoj;
4. Primera koncentriĝo estas kutime optimumigita inter 0.1uM kaj 1.0uM.
Elekto deROX:
En la procezo de kvanta reago, ROX povas ĝustigi la optikan vojon diferencon, pipet-eraron aŭ voluman diferencon kaŭzitan de vaporiĝo kaj kondensado unuforme, plibonigante la ripeteblon de la rezultoj.Tamen, oni devas rimarki, ke la elekto de ROX rilatas al la instrumento.Se la qPCR-instrumento havas la funkcion aŭtomate korekti la diferencon inter truoj, ĝi ne bezonas aldoni ROX;alie, ĝi bezonas aldoni ROX-korektadon.Malgrandaj partneroj en aĉetado de reakciiloj devas esti laŭ la instrumento uzata por elekti la ĝustan ROX, eviti postajn erarojn.
Preparado de reagsistemo:
Reakciaj volumoj de 20ul kaj 50ul estas preferitaj.La sekvaj aferoj devas esti atentitaj kiam la sistemo estas formulita:
1. La reagsistemo devas esti preparita per ventolado en la ultra-pura laborbenko, nova ddH2O estas uzata por ĉiu eksperimento;
2. Ĉiu eksperimento bezonas prepari NTC por kontroli ĉu estas poluo en la sistemo, kaj ĉiu paro da enkondukoj devas fari NTC kiam oni preparas la sistemon;
3. Por detekti ĉu ekzistas gDNA-restaĵo en la RNA-ŝablono, NRT povas esti preta por ĉiu specimeno por detekto;
4. Preparante la sistemon, oni rekomendas fari almenaŭ 3 teknikajn ripetojn por unu specimeno;
5. Kiam la ŝablono estas cDNA, oni rekomendas dilui 5-10 fojojn por redukti la inhibician efikon de inversa transskriba sistemo sur qPCR-eksperimento.Pli bone estas esplori la ŝablonkvanton laŭ gradiento, tiel ke la CT-valoro falas inter 20-30;
6. Determinu la bezonatan nombron da reagoj, pliigu je 5-10% surbaze de la nombro da reagoj, kaj kalkulu la voluman agordan nombron;
7, la sistemo estas preta uzante la premiksan principon, miksante post centrifugado kaj certigi neniujn vezikojn;
8, Laŭeble elekti subtenajn konsumaĵojn.
Rilata RT-qPCR-ilaro
La ilaro uzas unikan Foregene inversan transskriban reakciilon kaj Foregene HotStar Taq DNA Polymerase kombinitan kun unika reagsistemo por efike plibonigi la plifortigan efikecon kaj specifecon de la reago.
Afiŝtempo: Apr-23-2023