一、Pliigi la sentemon de la reagsistemo:
1. Izolu altkvalitan RNA:
Sukcesa cDNA-sintezo venas de altkvalita RNA.Altkvalita RNA devus almenaŭ esti plenlonga kaj libera de inversa transkriptazo-inhibitoroj kiel ekzemple EDTA aŭ SDS.La kvalito de RNA determinas la maksimuman kvanton de sekvencaj informoj, kiujn vi povas transskribi al cDNA.Ofta RNA-purigmetodo estas unu-paŝa metodo uzanta guanidinizotiocianato/acida fenolo.Por malhelpi poluadon de spurkvantoj de RNase, RNA izolita de RNase-riĉaj provaĵoj (kiel ekzemple pankreato) devas esti stokita en formaldehido por konservi altkvalitan RNA, precipe por longperspektiva stokado.La RNA eltirita de rathepato estis baze degradita post estado stokita en akvo dum unu semajno, dum la RNA eltirita de ratlieno restis stabila post estado stokita en akvo dum 3 jaroj.Krome, transskribaĵoj pli longaj ol 4 kb estas pli sentemaj al degenero de spuraj RNasoj ol malgrandaj transskribaĵoj.Por pliigi la stabilecon de stokitaj RNA-provaĵoj, RNA povas esti dissolvita en dejonigita formamido kaj stokita je -70 °C.Formamido uzita por konservi RNA devas esti libera de RNA-degradaj derompaĵoj.RNA de pankreato povas esti konservita en formamido dum almenaŭ unu jaro.Kiam vi preparas uzi RNA, vi povas uzi la jenan metodon por precipitar RNA: aldonu NaCl al 0.2M kaj 4 fojojn la volumeno de etanolo, metu ĉe ĉambra temperaturo dum 3-5 minutoj, kaj centrifugu je 10,000×g dum 5 minutoj.
2. Uzu la RNaseH-neaktivan (RNaseH-) inversan transskribazon:
RNase-inhibitoroj ofte estas aldonitaj al inversaj transskribaj reagoj por pliigi la longon kaj rendimenton de cDNA-sintezo.RNase-inhibitoroj devus esti aldonitaj dum la unuafadena sinteza reago en la ĉeesto de bufro kaj reduktanta agento (kiel ekzemple DTT), ĉar la proceso antaŭ cDNA-sintezo malnaturas la inhibitoron, tiel liberigante ligitan RNazon kiu povas degradi RNA.Proteinaj RNase-inhibidores nur malhelpas la degeneron de RNA fare de RNase A, B, C, kaj ne malhelpas RNazon sur la haŭto, do zorgu ne enkonduki RNazon el viaj fingroj malgraŭ la uzo de ĉi tiuj inhibidores.
Inversa transkriptazo katalizas la konvertiĝon de RNA al cDNA.Kaj M-MLV kaj AMV havas endogenan RNaseH-agadon aldone al sia propra polimeraza agado.RNaseH-agado kaj polimeraza agado konkuras kun unu la alian pri la hibrida fadeno formita inter la RNA-ŝablono kaj la DNA-enkonduko aŭ cDNA etendaĵofadeno, kaj degradas la RNA-fadenon en la RNA:DNA-komplekso.La RNA-ŝablono degradita per RNaseH-agado jam ne povas funkcii kiel efika substrato por cDNA-sintezo, kiu reduktas la rendimenton kaj longon de cDNA-sintezo.Tial, estus utile forigi aŭ multe redukti la RNaseH-agadon de inversa transkriptazo.。
SuperScript Ⅱ inversa transkriptazo, RNaseH- MMLV inversa transkriptazo kaj thermoScript inversa transkriptazo, RNaseH- AMV, povas akiri pli da kvanto kaj pli plenlonga cDNA ol MMLV kaj AMV.RT-PCR-sentemo estos tuŝita de la kvanto de cDNA-sintezo.ThermoScript estas multe pli sentema ol AMV.La grandeco de RT-PCR-produktoj estas limigita per la kapablo de inversa transkriptazo por sintezi cDNA, precipe dum klonado de pli grandaj cDNAoj.Kompare kun MMLV, SuperScripⅡ signife pliigis la rendimenton de longaj RT-PCR-produktoj.La RNaseH-inversa transskribazo ankaŭ havas pliigitan termostabilecon, tiel ke la reago povas esti farita ĉe temperaturoj pli altaj ol la normalaj 37-42 °C.Sub la proponitaj sintezaj kondiĉoj, uzu oligo(dT) enkondukon kaj 10 μCi de [α-P]dCTP.La totala rendimento de unua fadeno estis kalkulita per la TCA-pluvometodo.Plenlonga cDNA estis analizita uzante grand-orditajn bendojn eltranĉitaj kaj kalkulitaj je alkala agaroseĝelo.
3. Plialtigi la kobacian temperaturon por inversa transskribo:
Pli alta inkubacia temperaturo helpas malfermi la RNA-sekundaran strukturon, pliigante la rendimenton de la reago.Por la plej multaj RNA-ŝablonoj, kovado de la RNA kaj enkondukoj je 65 °C sen bufro aŭ salo, sekvita de rapida malvarmigo sur glacio eliminos la plej multajn sekundarajn strukturojn kaj permesos al enkondukoj ligi.Tamen, kelkaj ŝablonoj daŭre havas sekundarajn strukturojn, eĉ post varmecmalnaturado.Plifortigo de tiuj malfacilaj ŝablonoj povas esti farita uzante ThermoScript Reverse Transcriptase kaj metante la inversan transskriban reagon ĉe pli alta temperaturo por plibonigi plifortigon.Pli altaj kovadotemperaturoj ankaŭ povas pliigi specifecon, precipe kiam gen-specifaj enkondukoj (GSP) estas uzitaj por cDNA-sintezo (vidu Ĉapitro 3).Se vi uzas GSP, certigu, ke la Tm de la enkondukoj estas la sama kiel la atendata kova temperaturo.Ne uzu oligo(dT) kaj hazardajn enkondukojn super 60 °C.Hazardaj enkondukoj postulas kovadon je 25 °C dum 10 minutoj antaŭ pliiĝi al 60 °C.Aldone al uzado de pli alta inversa transskriba temperaturo, specifeco ankaŭ povas esti plibonigita rekte transdonante la RNA-/primermiksaĵon de la 65 °C denaturtemperaturo al la inversa transskriba inkubacia temperaturo kaj aldonante antaŭvarmigitan 2× reagmiksaĵon (cDNA varma-komenca sintezo).Tiu aliro helpas malhelpi la intermolekulan bazpariĝon kiu okazas ĉe pli malaltaj temperaturoj.La multobla temperaturŝanĝo postulata por RT-PCR povas esti simpligita uzante termociklilon.
Tth termostabila polimerazo funkcias kiel DNA-polimerazo en la ĉeesto de Mg2+ kaj kiel RNA-polimerazo en la ĉeesto de Mn2+.Ĝi povas esti varmigita je maksimuma temperaturo de 65 °C.Tamen, la ĉeesto de Mn2+ dum PCR reduktas fidelecon, kio igas Tth-polimerazon malpli taŭga por altprecizeca plifortigo, kiel ekzemple klonado de cDNA.Krome, Tth havas malaltan inversan transskriban efikecon, kiu reduktas sentemon, kaj, ĉar inversa transskribo kaj PCR povas esti faritaj per ununura enzimo, kontrolreagoj sen inversa transskribo ne povas esti uzitaj por kompari cDNA-plifortproduktojn kun poluado de genomic DNA.La plifortigaj produktoj estis apartigitaj.
4. Aldonaĵoj kiuj antaŭenigas inversan transskribon:
Aldonaĵoj inkluzive de glicerolo kaj DMSO estas aldonitaj al la unua-fadena sinteza reago, kiu povas redukti la stabilecon de la nuklea acido duoble-fadeno kaj malligi la sekundaran strukturon de RNA.Ĝis 20% glicerolo aŭ 10% DMSO povas esti aldonitaj sen influado de SuperScript II aŭ MMLV-agado.AMV ankaŭ povas toleri ĝis 20% de glicerolo sen perdo de agado.Por maksimumigi la sentemon de RT-PCR en la inversa transskriba reago de SuperScriptⅡ, 10% glicerolo povas esti aldonita kaj kovita je 45 °C.Se 1/10 de la reakcia reakcia produkto estas aldonita al PCR, tiam la koncentriĝo de glicerolo en la plifortiga reago estas 0,4%, kio ne sufiĉas por malhelpi PCR.
5. RNaseH-traktado:
Traktado de cDNA-sintezaj reagoj kun RNaseH antaŭ PCR povas pliigi sentemon.Por kelkaj ŝablonoj, supozeble ke RNA en la cDNA-sintezreago malhelpas la ligadon de plifortigproduktoj, en kiu kazo RNaseH-traktado povas pliigi sentemon.Ĝenerale, RNaseH-traktado estas necesa dum plifortigado de pli longaj plenlongaj cDNA-celŝablonoj, kiel ekzemple malalt-kopia tubera skerozo II.Por ĉi tiu malfacila ŝablono, RNaseH-traktado plibonigis la signalon produktitan de SuperScript II aŭ AMV-sintezita cDNA.Por la plej multaj RT-PCR-reagoj, RNaseH-traktado estas laŭvola, ĉar la PCR-malnaturaliĝa paŝo je 95 °C ĝenerale hidrolizas la RNA en la RNA:DNA komplekso.
6. Plibonigo de Malgranda RNA-Detekta Metodo:
RT-PCR estas aparte malfacila kiam nur malgrandaj kvantoj de RNA estas haveblaj.Glikogeno aldonita kiel portanto dum RNA-izolado helpas pliigi la rendimenton de malgrandaj provaĵoj.RNase-libera glikogeno povas esti aldonita samtempe kiel aldonado de Trizol.Glikogeno estas akvosolvebla kaj povas esti konservita en la akva fazo kun RNA por helpi postan precipitaĵon.Por specimenoj malpli ol 50 mg da histo aŭ 106 kulturitaj ĉeloj, la rekomendita koncentriĝo de RNase-libera glikogeno estas 250 μg/ml.
Aldonante acetiligitan BSA al la inversa transskriba reago uzante SuperScript II povas pliigi la sentemon, kaj por malgrandaj kvantoj de RNA, reduktante la kvanton de SuperScript II kaj aldoni 40 ekzemplerojn de RNaseOut-nukleazinhibitoro povas pliigi la nivelon de detekto.Se glikogeno estas uzita en la RNA-izolprocezo, estas ankoraŭ rekomendite aldoni BSA aŭ RNase-inhibitoron dum uzado de SuperScript II por inversa transskriba reago.
二、Pliigi RT-PCR-specifecon
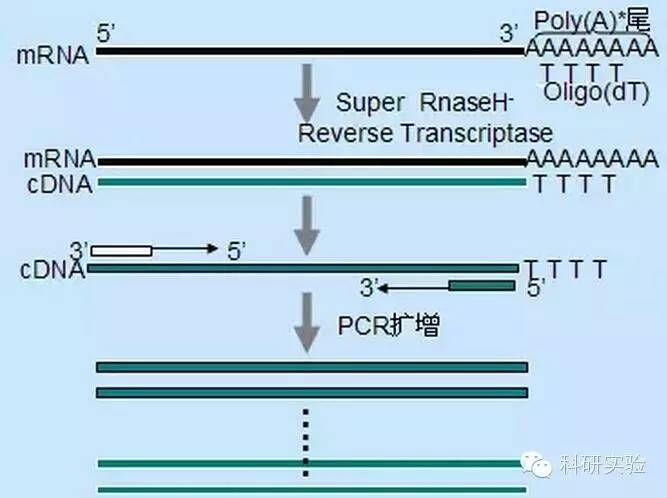
1. CND Asintezo:
Unufadena cDNA-sintezo povas esti iniciatita uzante tri malsamajn metodojn, kies relativa specifeco influas la kvanton kaj specon de cDNA sintezita.
La hazarda enkonduka metodo estis la malplej specifa el la tri metodoj.Enkondukoj kalmas ĉe multoblaj lokoj ĉie en la transskribaĵo, generante mallongajn, partlongajn cDNAojn.Tiu metodo estas ofte uzita por akiri 5′ finsekvencojn kaj por akiri cDNA de RNA-ŝablonoj kun regionoj de sekundara strukturo aŭ kun finlokoj kiuj ne povas esti reproduktitaj per inversa transkriptazo.Por akiri la plej longan cDNA, la rilatumo de enkondukoj al RNA en ĉiu RNA-provaĵo devas esti determinita empirie.La komenca koncentriĝo de hazardaj enkondukoj variis de 50 ĝis 250 ng per 20 μl reago.Ĉar cDNA sintezita de totala RNA uzanta hazardajn enkondukojn estas ĉefe ribosoma RNA, poli(A)+RNA estas ĝenerale elektita kiel la ŝablono.
Oligo (dT) enkondukoj estas pli specifaj ol hazardaj enkondukoj.Ĝi hibridiĝas al la poli(A) vosto trovita ĉe la 3′ fino de la plej multaj eŭkariotaj mRNA.Ĉar poli(A)+ RNA estas ĉirkaŭ 1% ĝis 2% de totala RNA, la kvanto kaj komplekseco de cDNA estas multe malpli ol kun hazardaj enkondukoj.Pro ĝia alta specifeco, oligo (dT) ĝenerale ne postulas optimumigon de la rilatumo de RNA al enkondukoj kaj poli (A)+ selektado.Oni rekomendas uzi 0.5μg oligo(dT) per 20μl reagsistemo.oligo(dT)12-18 taŭgas por plej multaj RT-PCR.La ThermoScript RT-PCR-Sistemo ofertas oligo(dT)20 pro sia pli bona termika stabileco por pli altaj kovadotemperaturoj.
Gen-specifaj enkondukoj (GSP) estas la plej specifaj enkondukoj por la inversa transskriba paŝo.GSP estas kontraŭsensa oligonukleotido kiu povas specife hibridigi al la RNA-celsekvenco, male al hazardaj enkondukoj aŭ oligo (dT), kiuj kalmas al ĉiuj RNAoj.La samaj reguloj uzitaj por dizajni PCR-enkondukojn validas por la dezajno de GSP en inverstransskribaj reagoj.La GSP povas esti la sama sekvenco kiel la plifortiga enkonduko kiu kalcinas al la 3′-plej fino de la mRNA, aŭ la GSP povas esti dizajnita por kalumi laŭflue de la inversa plifortiga enkonduko.Por kelkaj plifortigitaj temoj, pli ol unu kontraŭsensa enkonduko devas esti dizajnitaj por sukcesa RT-PCR ĉar la sekundara strukturo de la cela RNA povas malhelpi enkondukligadon.Oni rekomendas uzi 1 pmol kontraŭsensan GSP en 20 μl unuafadena sinteza reago.
2. Plialtigi la kobacian temperaturon por inversa transskribo:
Por plene utiligi la plenan avantaĝon de GSP-specifeco, inversa transkriptazo kun pli alta termostabileco devus esti uzita.Termostabilaj inversaj transkriptazoj povas esti kovataj ĉe pli altaj temperaturoj por pliigi reagostrecon.Ekzemple, se GSP kalmas je 55 °C, la specifeco de la GSP ne estos plene utiligita se AMV aŭ M-MLV estas uzitaj por inversa transskribo ĉe malalta severeco de 37 °C.Tamen, SuperScript II kaj ThermoScript povas esti reagita je 50 °C aŭ pli alta, kio eliminos nespecifajn produktojn generitajn ĉe pli malaltaj temperaturoj.Por maksimuma specifeco, la RNA/primermiksaĵo povas esti transdonita rekte de la 65 °C denatura temperaturo al la inversa transskriba kovado temperaturo kaj aldonita al antaŭvarmigita 2× reagmiksaĵo (cDNA-sintezo varma starto).Ĉi tio helpas malhelpi intermolekulajn bazpariĝon ĉe malaltaj temperaturoj.La multoblaj temperaturtransiroj postulataj por RT-PCR povas esti simpligitaj uzante termociklilon.
3. Reduktas genoman DNA-poluadon:
Ebla malfacileco renkontita kun RT-PCR estas la poluado de genoma DNA en la RNA.Uzante bonan RNA-izolan metodon, kiel Trizol Reagent, reduktos la kvanton de genoma DNA poluanta la RNA-preparon.Por eviti produktojn derivitajn de genoma DNA, RNA povas esti traktita kun plifortig-grada DNase I por forigi poluantan DNA antaŭ inversa transskribo.La DNase I-digestado estis finita per kovado de la specimenoj en 2.0 mM EDTA dum 10 minutoj je 65 °C.EDTA povas kelati magneziojonojn, malhelpante magneziojon-dependan RNA-hidrolizon ĉe altaj temperaturoj.
Por apartigi plifortigitan cDNA de poluado de genomic-DNA plifortigproduktoj, enkondukoj povas esti dizajnitaj ke ĉiu anela por apartigi eksonojn.PCR-produktoj derivitaj de cDNA estos pli mallongaj ol tiuj derivitaj de poluita genoma DNA.Krome, kontroleksperimento sen inversa transskribo estis farita sur ĉiu RNA-ŝablono por determini ĉu antaŭfiksita fragmento estis derivita de genoma DNA aŭ cDNA.La PCR-produkto akirita sen inversa transskribo estas derivita de la genaro.
Afiŝtempo: majo-16-2023








