Komenca materialo: RNA
Kvanta inversa transskriba PCR (RT-qPCR) estas eksperimenta metodo uzita en PCR-eksperimentoj utiligantaj RNA kiel la startmaterialon.En tiu metodo, totala RNA aŭ mesaĝa RNA (mRNA) unue estas transskribitaj en komplementan DNA (cDNA) per inversa transkriptazo.Poste, qPCR-reago estis farita uzante la cDNA kiel ŝablonon.RT-qPCR estis uzita en gamo da molekula biologiaj aplikoj, inkluzive de genekspresio-analizo, RNA-interfervalidumado, mikroarta validumado, patogendetekto, genetika testado, kaj malsanesplorado.
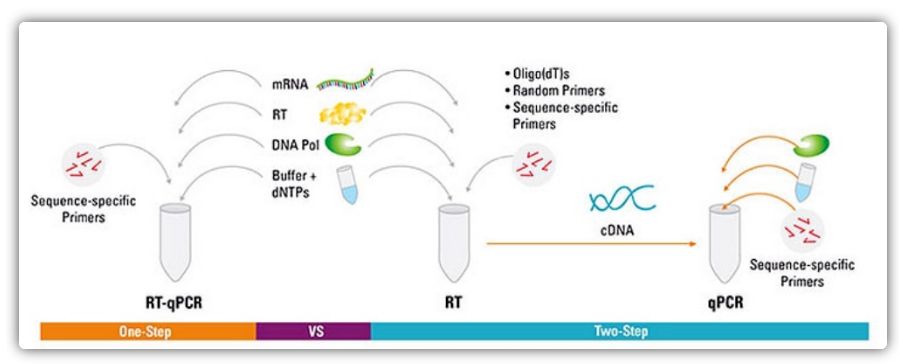
Unu-paŝaj kaj du-paŝaj metodoj por RT-qPCR
RT-qPCR povas esti plenumita per unu-paŝa aŭ du-paŝa metodo.Unu-paŝa RT-qPCR kombinas inversan transskribon kaj PCR-plifortigon, permesante inversan transkriptazon kaj DNA-polimerazon kompletigi la reagon en la sama tubo sub la samaj bufrokondiĉoj.Unu-paŝa RT-qPCR nur postulas la uzon de sekvenc-specifaj enkondukoj.En du-ŝtupa RT-qPCR, inversa transskribo kaj PCR-plifortigo estas faritaj en du tuboj, uzante malsamajn optimumigitajn bufrojn, reagkondiĉojn, kaj komencajn dezajnostrategiojn.
| Avantaĝo | Malavantaĝo | |
| Unu Paŝo | Tiu metodo havas malpli eksperimentan eraron ĉar ambaŭ reagoj estas faritaj en unu tubo
Malpli da pipetaj paŝoj reduktas la riskon de poluado
Taŭga por alt-trafia plifortigo/ekstruado, rapida kaj reproduktebla | Du-ŝtupaj reagoj ne povas esti optimumigitaj aparte
Ĉar la reagkondiĉoj estas endanĝerigitaj kombinante la dupaŝan reakcion, la sentemo ne estas same bona kiel tiu de la dupaŝa metodo
La nombro da celoj detektitaj per ununura provaĵo estas malgranda |
| Du Paŝoj | Kapablo krei stabilajn cDNA-bibliotekojn kiuj povas esti stokitaj dum longaj tempodaŭroj kaj uzataj en multoblaj reagoj
Celgenoj kaj referencgenoj povas esti plifortigitaj de la sama cDNA-biblioteko sen la bezono de multoblaj cDNA-bibliotekoj
Reagbufroj kaj reakcikondiĉoj kiuj ebligas optimumigon de ununuraj reagkuroj
Fleksebla elekto de ellasilkondiĉoj | Uzado de multoblaj tuboj kaj pli da pipetaj paŝoj pliigas la riskon de DNA-poluado, kaj tempopostula.
Postulas pli da optimumigo ol la unupaŝa metodo |
Rilataj produktoj:
RT-qPCR Easyᵀᴹ (Unu Paŝo) - SYBR Verda I
RT-qPCR Easyᵀᴹ (Unu Paŝo) - Taqman
RT Easyᵀᴹ I Master Premix For First-Strand CDNA-Sintezo
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Selektado de totala RNA kaj mRNA
Dum dizajnado de RT-qPCR-eksperimento, estas grave decidi ĉu uzi totalan RNA aŭ purigitan mRNA kiel ŝablonon por inversa transskribo.Kvankam mRNA eble povas disponigi iomete pli altan sentemon, totala RNA daŭre estas ofte uzita.La kialo de tio estas ke totala RNA havas pli gravan avantaĝon kiel startmaterialo ol mRNA.Unue, la procezo postulas malpli da purigaj paŝoj, kio certigas pli bonan kvantan reakiron de ŝablono kaj pli bonan normaligon de rezultoj al komencaj ĉelaj nombroj.Due, ĝi evitas la mRNA-riĉigan paŝon, kiu povas eviti la eblecon de misformitaj rezultoj pro malsamaj reakiroj de malsamaj mRNAoj.Entute, ĉar en la plej multaj aplikoj la relativa kvantigo de la celgeno estas pli grava ol la absoluta sentemo de la detekto, totala RNA estas pli taŭga en la plej multaj kazoj.
Inversa transskriba enkonduko
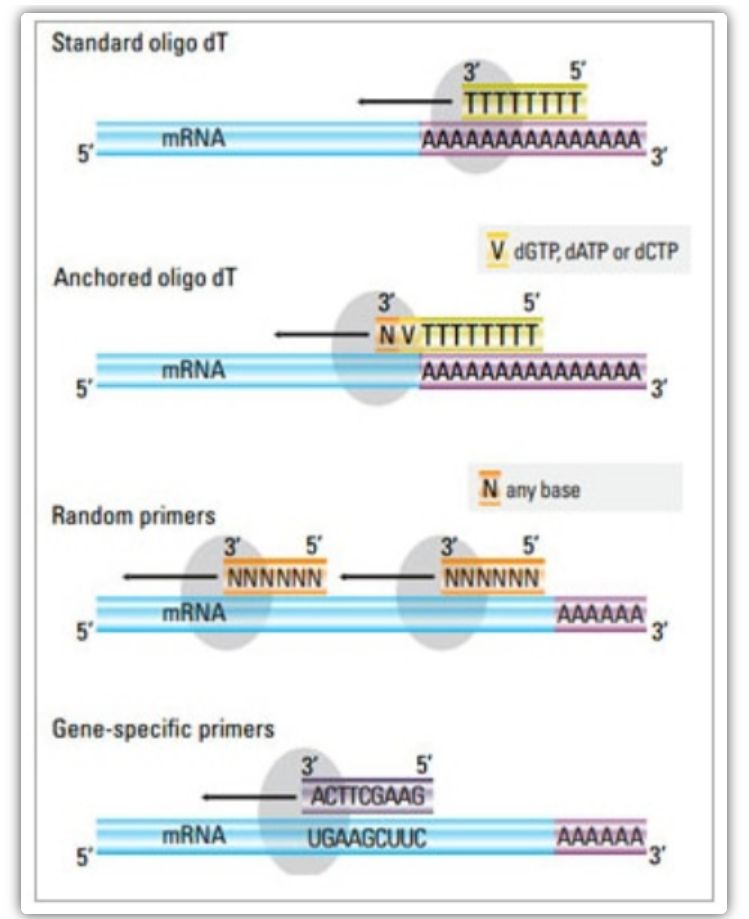
En la du-ŝtupa metodo, tri malsamaj metodoj povas esti uzitaj por prepari la cDNA-reagon: oligo (dT) enkondukoj, hazardaj enkondukoj, aŭ sekvenc-specifaj enkondukoj.Tipe, oligo (dT) enkondukoj kaj hazardaj enkondukoj estas uzitaj en kombinaĵo.Tiuj enkondukoj kalmas al la ŝablonmRNA-fadeno kaj disponigas inversan transkriptazon kun deirpunkto por sintezo.
| Unua elekto | Strukturo kaj funkcio | Avantaĝo | Malavantaĝo |
| Oligo (dT) enkonduko (aŭ ankrita oligo (dT) enkonduko) | Plilongigita kalciado al timinrestaĵoj ĉe la poli(A) vosto de mRNA;ankro-oligo (dT) enkonduko enhavas G, C, aŭ A ĉe la 3′ fino (ankroloko) | Sintezo de plenlonga cDNA de poli(A)-vosta mRNA
Aplika kiam malpli da komenca materialo estas disponebla
Ankri ejo certigas ke la oligo (dT) enkonduko ligas al la 5′ poli (A) vosto de la mRNA | Taŭga nur por plifortigi genojn kun poli(A) vostoj
Akiru cDNA detranĉitan de la primara loko*2 en poli(A)
Partia ligi al la 3′ fino*
*Ĉi tiu ebleco estas minimumigita se ankritaj oligo(dT) enkondukoj estas uzitaj |
| hazarda enkonduko
| 6 ĝis 9 bazoj en longo, kiuj povas kalumi al multoblaj ejoj dum RNA-transskribo | Anneal al ĉiuj RNAoj (tRNA, rRNA, kaj mRNA)
Taŭga por transskribaĵoj kun signifa sekundara strukturo, aŭ kiam malpli komenca materialo estas havebla
Alta cDNA Rendimento | cDNA estas inversa transskribita de ĉiu RNA, kiu kutime ne estas dezirata kaj povas dilui la signalon de la celmRNA.
akiri detranĉitan cDNA |
| sekvenco-specifaj enkondukoj | Propraj enkondukoj celantaj specifajn mRNA-sekvencojn | specifa cDNA biblioteko
Plibonigi sentemon
Uzante inversajn qPCR-komencojn | Nur limigita al la sintezo de ununura celgeno |
Inversa transkriptazo
Inversa transkriptazo estas enzimo kiu uzas RNA por sintezi DNA.Kelkaj inversaj transkriptazoj havas RNazaktivecon kaj povas degradi RNA-fadenojn en RNA-DNA hibridaj fadenoj post transskribo.Se ĝi ne havas RNase-enzimecan agadon, RNaseH povas esti aldonita por pli alta qPCR-efikeco.Ofte uzitaj enzimoj inkludas Moloney murine leŭkemiovirusan inversan transkriptazon kaj birdan mieloblastomvirusan inversan transkriptazon.Por RT-qPCR, estas ideale elekti inversan transkriptazon kun pli alta termostabileco, tiel ke cDNA-sintezo povas esti farita ĉe pli altaj temperaturoj, certigante sukcesan transskribon de RNAoj kun pli alta sekundara strukturo, konservante ilian plenan agadon dum la reago, rezultigante pli altajn cDNA-rendimentojn.
Rilataj produktoj:
Foreasy M-MLV Reverse Transcriptase
RNase H-agado de inversa transkriptazo
RNaseH povas degradi RNA-fadenojn de RNA-DNA-dupleksoj, permesante efikan sintezon de duoble-senhelpa DNA.Tamen, dum uzado de longa mRNA kiel ŝablono, la RNA povas esti degradita trofrue, rezultigante stumpigitan cDNA.Tial, estas ofte utile minimumigi RNaseH-agadon dum cDNA-klonado se sintezo de longaj transskribaĵoj estas dezirata.En kontrasto, inversaj transkriptazoj kun RNase H-agado ofte estas utilaj por qPCR-aplikoj ĉar ili plifortigas la fandadon de RNA-DNA-dupleksoj dum la unua ciklo de PCR.
Unua dezajno
PCR-enkondukoj uzitaj por la qPCR-paŝo en RT-qPCR devus ideale esti dizajnitaj por enhavi ekson-eksonan krucvojon, kie plifortiga enkonduko eble povus enhavi faktan ekson-intronan limon.Ĉar intron-enhavantaj genomic DNA-sekvencoj ne estas plifortigitaj, tiu dezajno reduktas la riskon de falsaj pozitivoj plifortigitaj de poluado de genomic DNA.
Se enkondukoj ne povas esti dizajnitaj por apartigi ekson aŭ ekson-ekson-limojn, povas esti necese trakti RNA-provaĵojn kun RNase-libera DNase I aŭ dsDNase por forigi genoman DNA-poluadon.
RT-qPCR-kontrolo
Inversa transskriba negativa kontrolo (-RT-kontrolo) devus esti inkludita en ĉiuj RT-qPCR-eksperimentoj por detekti DNA-poluadon (kiel ekzemple genoma DNA aŭ PCR-produktoj de antaŭaj reagoj).Tiu kontrolo enhavas ĉiujn reagkomponentojn krom inversa transkriptazo.Ĉar inversa transskribo ne okazas kun tiu kontrolo, se PCR-plifortigo estas observita, poluado de DNA estas plej verŝajna.
Afiŝtempo: Aŭg-02-2022








